控制基因敲除法
控制基因敲除法
【探针技术用于检测由siRNA介导的基因调制】
为了研究基因功能,科学家们常常会有针对性地关断某些特定的基因。 而要验证这种基因沉默的有效性, 就需要运用适当的具特异性的检测方法。理想的检测方法不仅要测量准确, 而且还要能让细胞保持完好无损。
利用诸如RNA(核糖核酸)技术来调节基因表达已成为研究基因功能和生物反应机理的一种基本手段。常规的测定基因沉默率(基因敲除)的方法,需要采用细胞裂解或细胞壁通透以及细胞固定的方法将细胞加以破坏。而且这些技术的缺点还在于所测定的结果只能反映细胞群体的平均的基因表达。
转染报告构建是一种对细胞无损害的检测方法。然而这种方法虽能让细胞保持活性, 却不能对内源基因的表达进行检验。此外, 该方法对细胞的活性也有负面影响。理想的、非破坏性的RNA检测试剂不仅要适合基因表达的研究, 还应能够用于后续离析得到的活细胞的分选及分析中。
Smartflare“灵光”RNA检测探针(EMD 密理博公司, 比尔里卡/美国)能够检测在活细胞中的目标RNA和微RNA水平。该探针不需载体就可进入细胞中, 且对细胞的活性无影响。此技术使得快速验证基因表达成为可能。而且可结合应用细胞类型的分选手段, 可分选出所需的细胞群。此外, 探针试剂不需要样品制备, 细胞经过检测过程后不被破坏, 可在后续分析中继续使用。
事实证明, 应用针对选定的目标RNA有特异性的探针,实现了准确有效地检测siRNA介导的基因敲除。这种独创的检测单个细胞的RNA水平的探针技术,为实现生物路径或生理过程与基因功能这两个研究领域之间的联系提供了新的可能性。
基因敲除和检测:材料和方法
本文中所涉及的基因敲除, 流式细胞仪的mRNA(信使核糖核酸)测定和qRT-PCR(实时定量聚合酶链式反应)作为对比检测的操作过程如下进行:
• 基因敲除:将SCC12细胞(人体鳞状细胞癌)和LNCaP细胞(人类前列腺癌)接种于96孔微量滴定板上(10000个细胞/孔)。放置24小时后, 用对照siRNA及对生存素有特异性的siRNA来进行细胞转染并孵育48小时。存活素是一种细胞凋亡抑制剂蛋白, 在许多癌细胞系中有明显的升高。 孵育完成后, 更新培养基以去除siRNA和转染试剂。
• 调制检测:经过siRNA处理后的SCC12和LNCaP细胞,加上对生存素具特异性的Smartflare“星光”探针或Smartflare“星光”对照探针的1000倍稀释液,一并在细胞培养基里孵育过夜。于次日将细胞用胰蛋白酶处理, 并用流式细胞仪(Guava EasyCyte 8HT)对其进行分析。同时, 将那些不作为采用Smartflare“星光”探针进行进一步研究的、业已经过siRNA处理过的细胞收集起来,用于进行实时定量逆转录聚合酶链式反应 (qRT-PCR),作为对比定量分析之用。
• qRT-PCR:RNA总量用RNeasy试剂包(Qiagen公司)进行萃取, 然后加入到Taqman RNA-to-Ct1-Step Kit 试剂包(LifeTechnologies)的溶液中。实时定量聚合酶链式反应采用LightCycler 480系统 (Roche公司)进行。
mRNA的检测分析结果
本研究中获得了如下的分析结果:
•流式细胞仪测定mRNA: 为检测基因敲除,将存活素特异性Smartflare“星光”探针和对照探针与用存活素和对照siRNA处理过的SCC12和LNCaP细胞一起孵育。特异性探针处理过的不同细胞的目标基因,其表达水平的差异通过流式细胞仪分析测定(见图2)。
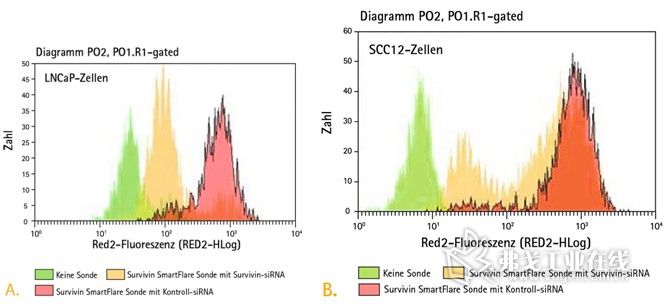
图2.通过测定在LNCaP和SCC12细胞中存活素基因敲除的相对Smartflare“星光”信号,可以显示其间的区别。直方图中的 Smartflare“星光”信号,分别对应于具有或没有进行存活素的siRNA敲除情况下,存活素在LNCaP细胞中(A)和SCC12细胞(B)中的 表达。
• 借助于qRT-PCR进行mRNA的检测:为了利用qRT-PCR进行基因敲除的检测, 将存活素特异性Smartflare“星光”探针和控制探针,与事先通过存活素和控制siRNA处理过的SCC12细胞和LNCaP细胞一起进行孵育。由Smartflare“星光”探针技术所测得的相对基因表达水平 采用柱状图表示,并与由qRT-PCR所得到的数据进行比较表明, 这种相对的基因表达水平在量化方面是相似的,而与敲除技术的测量无关(见图3)。
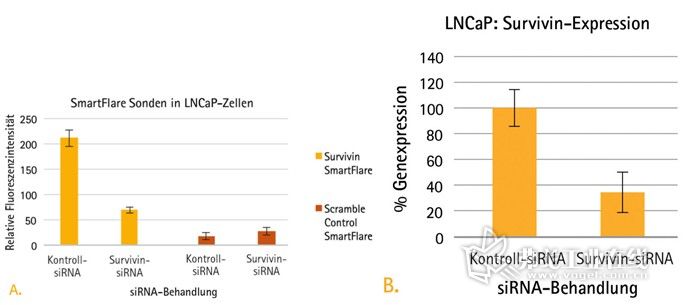
图3.在LNCaP和SCC12细胞内通过RNAi进行的存活素基因敲除,可以通过Smartflare“星光”探针和qRT-PCR的检测加以区别。
(A) 通过Smartflare“星光”探针技术和流式细胞仪分析测定,可以得到在具有或没有存活素敲除的情况下的LNCaP细胞中存活素表达。
(B) 通过qRT-PCR测定,求证了LNCaP细胞中有和没有存活素敲除情况,存活素的相对表达水平。
由于Smartflare“星光”探针技术检测的是单一的无损细胞中的基因表达,由此我们可以获取各种经不同方式处理过的细胞的分布信息。在此我们特别观察到,受存活素特异性Smartflare“星光”探头处理过的LNCaP细胞的生存信号具有单峰式分布,而在SCC12细胞中是双峰分布。
单个活细胞的胞内基因表达
研究结果表明, Smartflare“星光”探针技术可以检测单个活细胞的胞内基因表达。这种能力对涉及RNAi(RNA干扰)介导的基因敲除的研究至关重要, 尤其是在还未确定不完整敲除原因的情况下。由于使用这种探针技术可以检测单个细胞的基因敲除的不同程度,以得到关于基因表达的细胞与细胞间的演变、基因敲除和siRNA的渗透效率的信息。这些信息对解释通过RNAi处理后所得到的分析结果非常有用。此外, 探针技术还允许实现细胞的分选和培养回收, 从而使得相同的细胞样本能够再用于后续分析, 诸如抗体染色, 流式细胞仪和qRT-PCR检测等。可以说,测量基因敲除后的同种单个细胞样品内的生理变化成为可能, 这对于考察基因表达和细胞表型之间的相关性来说有重要意义。
细胞内的基因表达
在研究通过RNAi介导的基因敲除时,难点在于找出某种不完全敲除根源的所在。尤其是传统RNA定量检测(测的是平均RNA水平),无法区分基因敲除失效的原因,是因siRNA序列的设计不良?还是因siRNA未渗透到目标细胞,?还是由于目标细胞内的内源性基因表达差异太大?因此,进行单个活细胞的胞内基因表达的检测就有着决定性的意义。
访谈:活细胞中的RNA分析

图4.Eva Trisch博士, 默克密理博技术销售专家
LP:Eva Trisch女士, 新的RNA检测探针技术是如何构建的 ? 用于哪些方面?
Eva Trisch博士:Smartflare “星光”RNA检测探针是在水中重建而成, 被赋予了细胞培养基, 并用细胞孵育过夜。在此过程中探针通过自然吸收机制的内吞作用而进入到细胞中。一个Smartflare“星光”探头由一个金纳米粒子与数个“捕获链”耦合而成。这些DNA链的长度为40个核苷酸, 其序列与目标RNA序列的一部分互补。捕捉链存载着与其互补的“报告链”。也就是说此捕捉链载有目标RNA序列的一部分。在报告链上耦合了一个荧光集团(Cy3或y5),这些荧光集团靠近金颗粒末端。
LP: 怎样进行单细胞基础上的RNA水平的特异性检测 ?
Trisch:由于荧光集团与金颗粒貼近而金有着荧光淬灭剂的属性, 所以探针处于基态时没有荧光信号发射。在胞质溶胶中若有目标RNA存在, 这些目标RNA通过序列互补被结合到捕获链并将报告链解放出来, 这是由于内源目标RNA对捕获链的亲和力高于报告链,这是一个竞争反应。由于报告链离开了金颗粒, 荧光淬灭被解除, 从而胞质溶胶中出现了可以检测到的荧光信号。
LP: 具体说来这种技术会带来哪些新的机遇 ?
Trisch:Smartflare“星光”探头可以测定单个活细胞的RNA水平而不损坏细胞,也不影响基因表达,细胞保持活性并且可进一步用于下游分析。研究者检测到单个细胞内的目标RNA的荧光值(非总体分布的平均值), 可以表征细胞群的分布。而亚群分布可以在流式细胞仪设备上得以分选。
LP:怎么能确保细胞在被检测时不会受到损害 ?
Trisch:金纳米粒子不需转染试剂来进入细胞,并对细胞无毒性。它们通过内吞作用进入到细胞, 使细胞得以保存。因此细胞存活研究可证明, 加入或不加Smartflare“星光”探针的情况下细胞的存活状况是相同的。
LP:新的检测探针无需样品制备,用户在使用时应注意什么 ?
Trisch:用户得到的样品是冻干产物, 在4℃下可保持五年。在50µl水中重组后探头应避光并且室温下(非冷藏)保存。
海姆霍兹环保研究中心
展源
何发
相关文章
-

三聚氰胺分析仪快速筛查奶制品
2020-05-27
-

三聚氰胺分析仪快速筛查奶制品
2020-05-27
-

胶囊中的铬的快速筛查
2020-05-27
-

代谢产物的筛查和鉴定
2023-03-03
-

磨碎筛分设备的新应用
2020-05-27
-

肉类中抗生素残留筛查方案
2020-05-27
-

基因疗法再出击
2020-05-27
-

基因疗法再出击
2020-05-27
-

肉产品中抗生素残留筛查方案
2020-05-27

加载更多